Capitolo 18 Richiami sul modello lineare (LM)
18.1 Esempio: Regressione per effetto tranquillante su topi
18.1.1 Descrizione dei dati
In un esperimento in cui si investiga l’effetto di un tranquillante, sono misurati i tempi di reazione ad uno stimolo di topi dopo che a ciascuno di questi è stato somministrata una dose diversa del farmaco.
18.1.2 Caricamento dei dati
## Classes 'tbl_df', 'tbl' and 'data.frame': 10 obs. of 3 variables:
## $ rat : int 1 2 3 4 5 6 7 8 9 10
## $ dose: num 0 0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9
## $ time: num 0.32 0.24 0.4 0.52 0.44 0.56 0.64 0.52 0.6 0.8## # A tibble: 6 x 3
## rat dose time
## <int> <dbl> <dbl>
## 1 1 0 0.32
## 2 2 0.1 0.24
## 3 3 0.2 0.4
## 4 4 0.3 0.52
## 5 5 0.4 0.44
## 6 6 0.5 0.5618.1.3 Statistiche descrittive
Poiché siamo principalmente interessati alla relazione tra la dose del farmaco e i tempi di reazione (time),
iniziamo una analisi descrittive tracciando un grafico che mette in relazione queste due variabili.
ggp <- ggplot(data=drug, mapping = aes(x=dose, y=time)) +
geom_point(color="darkblue", size=2) +
geom_smooth(method = "lm", colour="red", se = FALSE) +
geom_smooth(method = "loess", colour="green", se = FALSE, span=1) +
xlab("dose (mg)") + ylab("Tempo di reazione (sec)")
print(ggp)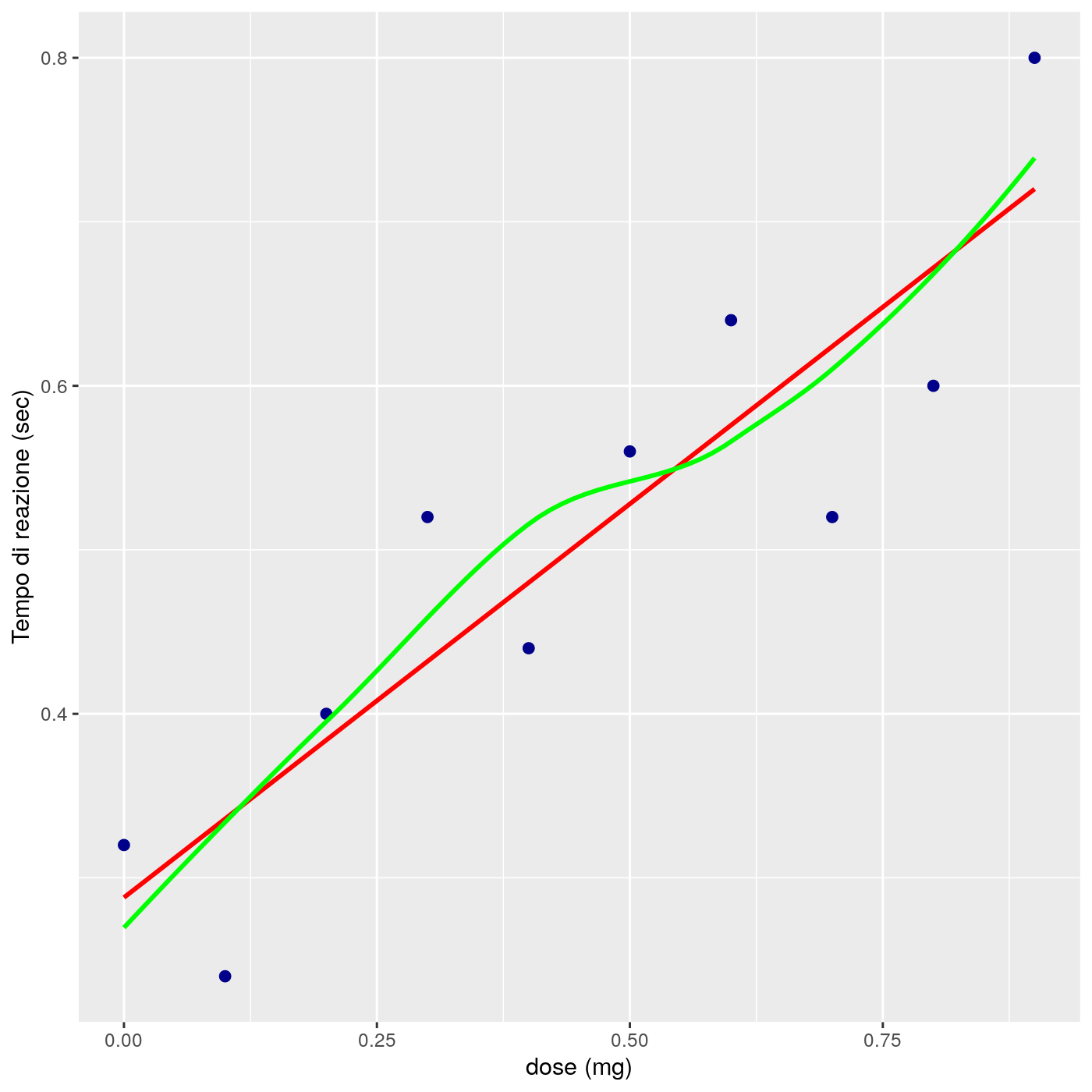
Figura 18.1: Scatterplot di tempo Vs. dose con regressione lineare e linea loess
Nel codice qui sopra, method="lm" della chiamata della funzione geom_smooth()
aggiunge la retta di regressione all’oggetto ggp e method="loess" di geom_smooth()
aggiunge una linea di regressione locale all’oggetto ggp.
Probabilmente una retta di regressione semplice è una buona scelta per descrivere la relazione.
18.1.4 Inferenza e modelli
Adattiamo quindi un semplice modello lineare \(E(y_i \vert xi) = \beta_0 + \beta_1 \cdot x_i\) tra il tempo di reazione e la dose del farmaco e produciamo un riepilogo del modello adattato.
##
## Call:
## lm(formula = time ~ dose, data = drug)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.104 -0.064 0.024 0.056 0.088
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.28800 0.04522 6.368 0.000216 ***
## dose 0.48000 0.08471 5.666 0.000472 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.07694 on 8 degrees of freedom
## Multiple R-squared: 0.8005, Adjusted R-squared: 0.7756
## F-statistic: 32.11 on 1 and 8 DF, p-value: 0.0004724Leggendo i risultati, sembra che aumentando la dose di una unità, il tempo (time) cresce in media
di 0.48, e questa crescita sia significativa.
Un altro modo per ottenere il p-value per il parametro di dose è confrontare il modello time ~ dose
col modello time ~ 1, e testare quindi la significatività del coefficiente angolare tramite anova()
## Analysis of Variance Table
##
## Model 1: time ~ dose
## Model 2: time ~ 1
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 8 0.04736
## 2 9 0.23744 -1 -0.19008 32.108 0.0004724 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Il risultato è lo stesso.
18.1.5 Analisi dei residui
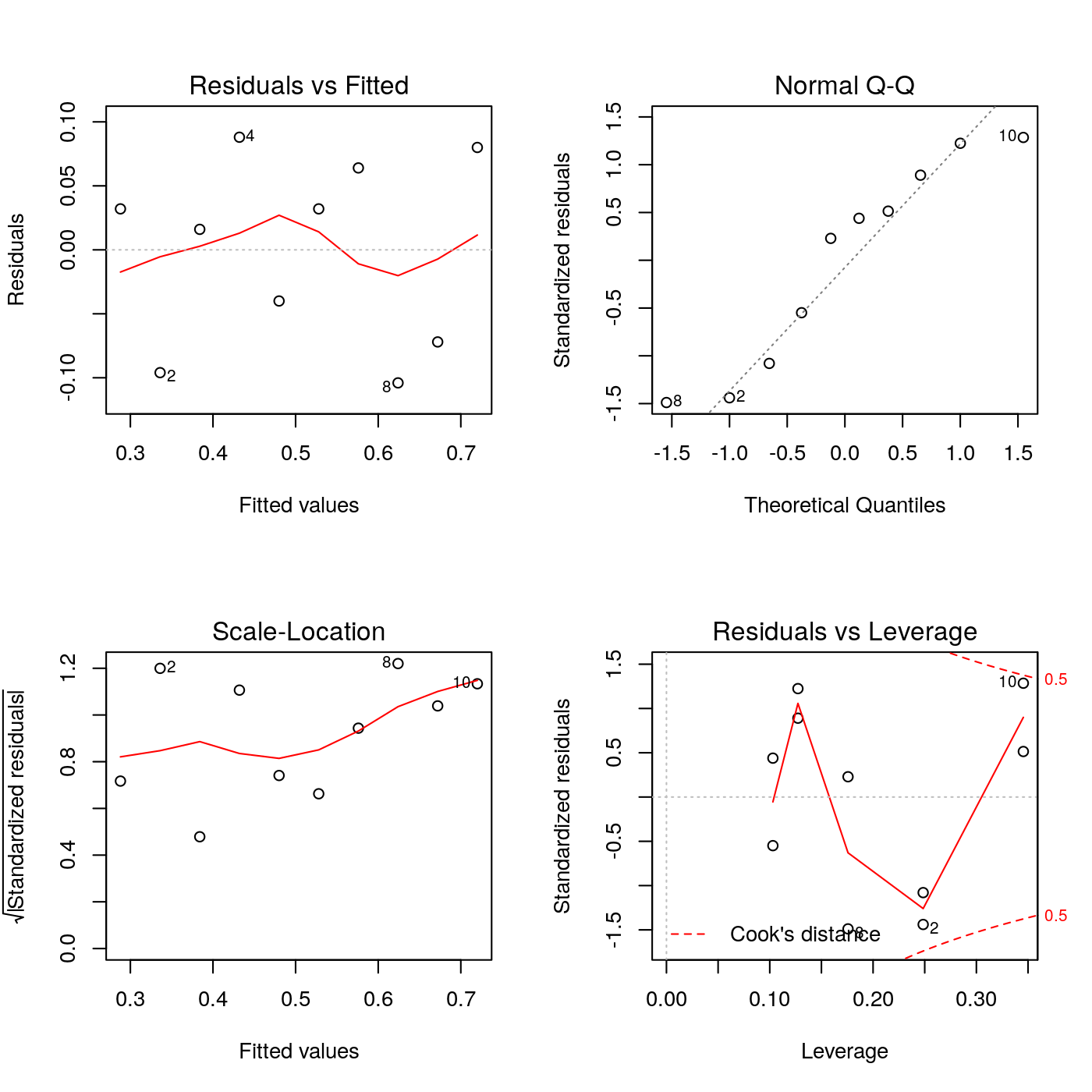
Figura 18.2: Grafico dei residui del modello lineare semplice
I grafici non evidenziano problemi particolari.
18.2 Esempio: Regressione su poliesterificazione
18.2.1 Descrizione dei dati
Nello studio della poliesterificazione degli acidi grassi con glicoli, è studiato
l’effetto della temperatura (temperature \(^◦\)C) sulla percentuale di conversione del processo
di esterificazione (conversion). I dati sono il risultato di un esperimento che usa un catalizzatore
di \(4 \cdot 10^{−4}\) mole zinc chloride ogni 100 grammi di acidi grassi.
18.2.2 Caricamento dei dati
## Classes 'tbl_df', 'tbl' and 'data.frame': 6 obs. of 2 variables:
## $ temperature: int 175 200 225 250 275 300
## $ conversion : int 67 83 93 97 99 92## # A tibble: 6 x 2
## temperature conversion
## <int> <int>
## 1 175 67
## 2 200 83
## 3 225 93
## 4 250 97
## 5 275 99
## 6 300 9218.2.3 Statistiche descrittive
Rappresentiamo graficamente la relazione tra la variabile dipendente e la variabile indipendente.
ggp <- ggplot(data = polyester, mapping=aes(x = temperature, y=conversion)) +
geom_point(color="darkblue") + xlab("temperatura (°C)") +
ylab("Percentuale di conversione (%)")
print(ggp)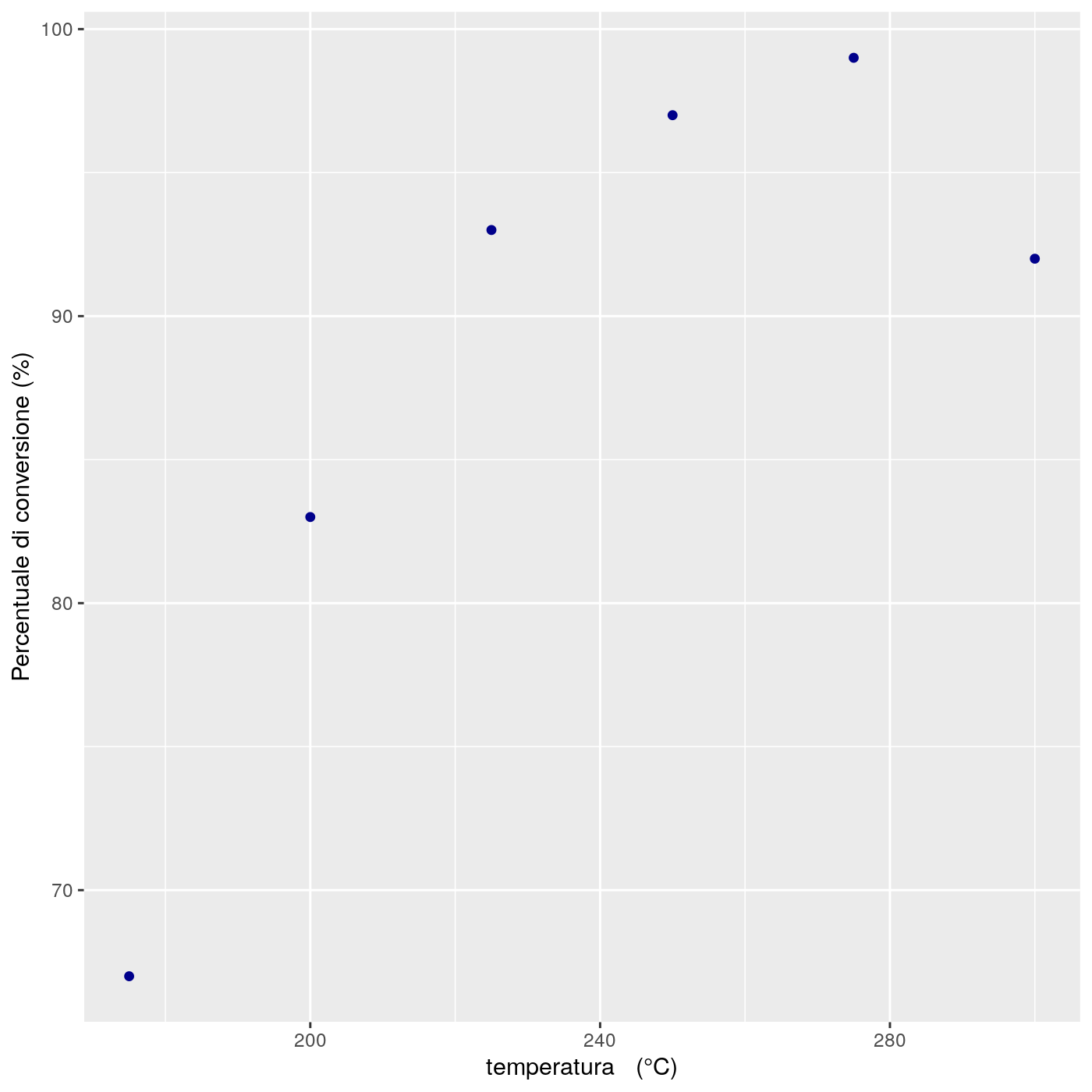
Figura 18.3: Scatterplot della conversione Vs. temperatura
Sembra che una funzione quadratica (una parabola) possa descrivere bene i dati.
18.2.4 Inferenza e modelli
A dispetto dell’evidenza qui sopra, proviamo inizialmente a stimare un modello lineare semplice, e produrre un grafico illustrativo del modello stesso.
##
## Call:
## lm(formula = conversion ~ temperature, data = polyester)
##
## Residuals:
## 1 2 3 4 5 6
## -8.857 2.086 7.029 5.971 2.914 -9.143
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 40.45714 18.60031 2.175 0.0953 .
## temperature 0.20229 0.07708 2.624 0.0585 .
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 8.061 on 4 degrees of freedom
## Multiple R-squared: 0.6326, Adjusted R-squared: 0.5407
## F-statistic: 6.887 on 1 and 4 DF, p-value: 0.05853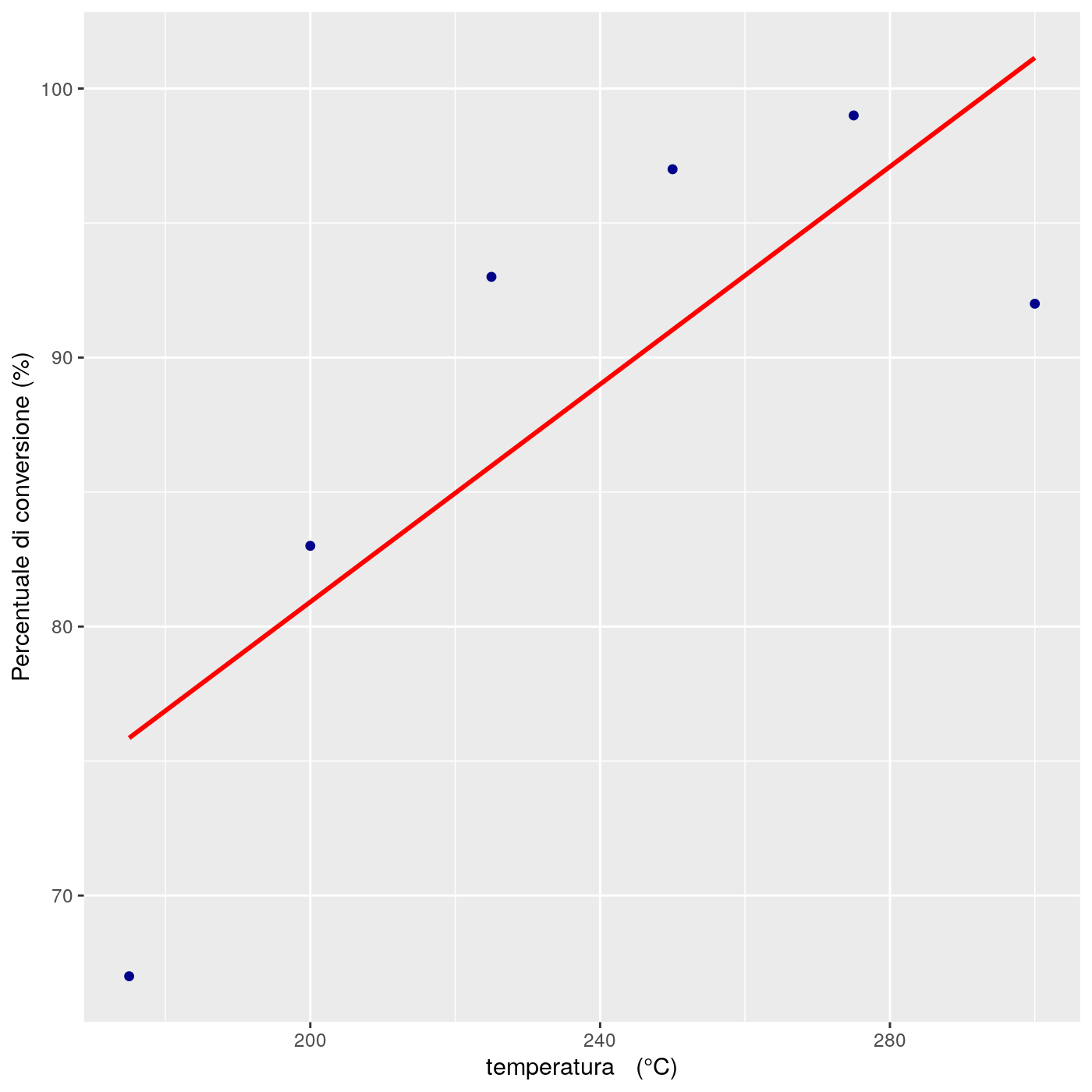
Figura 18.4: Scatterplot di conversione Vs. temperatura con modello lineare
Ora il grafico dei residui.
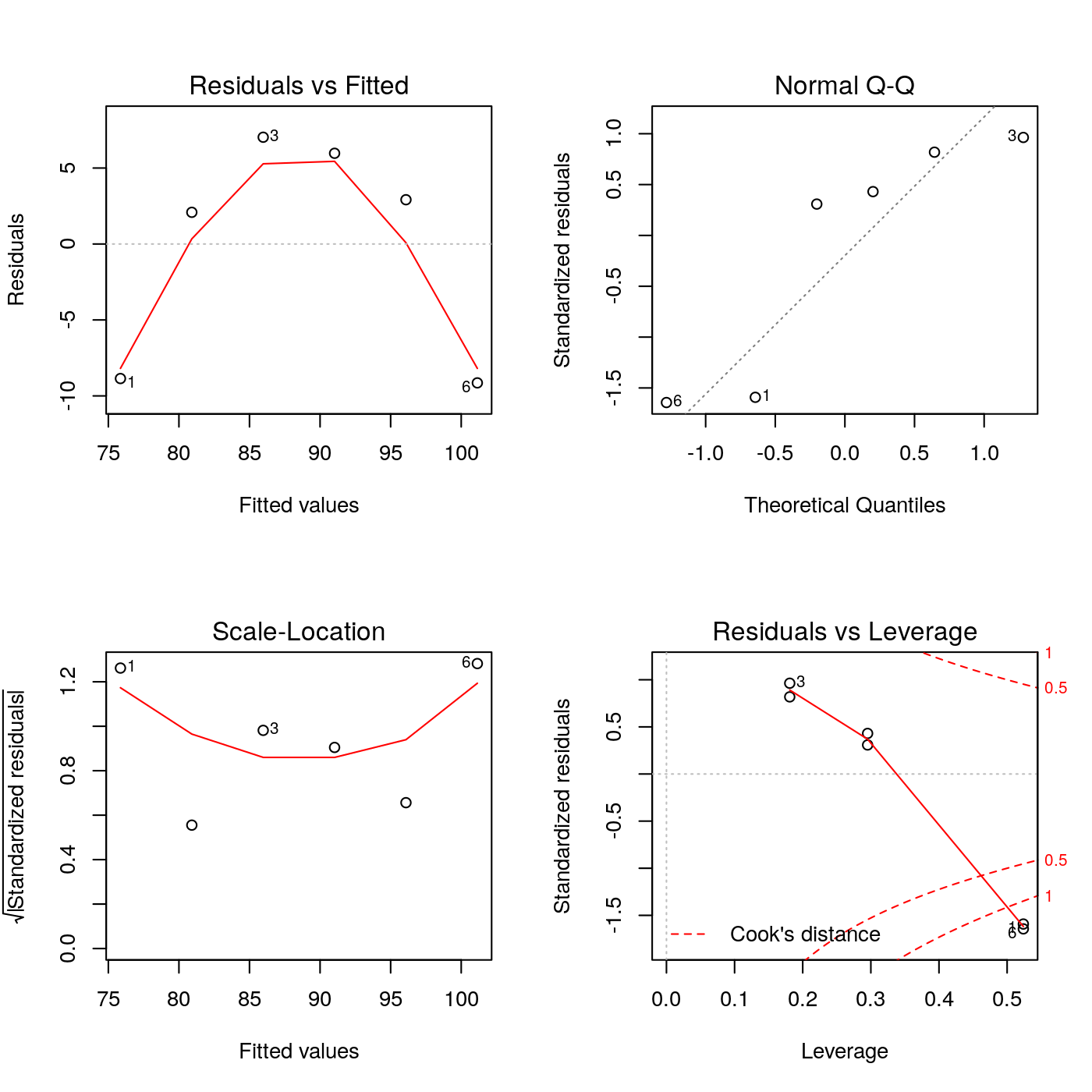
Figura 18.5: Analisi dei residui del modello lineare semplice conversione Vs. temperatura
Entrambi i grafici suggeriscono che potrebbe esistere un effetto di curvatura.
Un atro grafico per verificare questa ipotesi, è il grafico dei residui Vs.
la variabile indiependente:
df <- data.frame(temperature = polyester$temperature, mod_residuals =residuals(fm0))
ggp <- ggplot(data = df, mapping = aes(x=temperature, y=mod_residuals)) +
geom_point(color="darkblue") + geom_hline(yintercept = 0, colour="darkgreen")
print(ggp)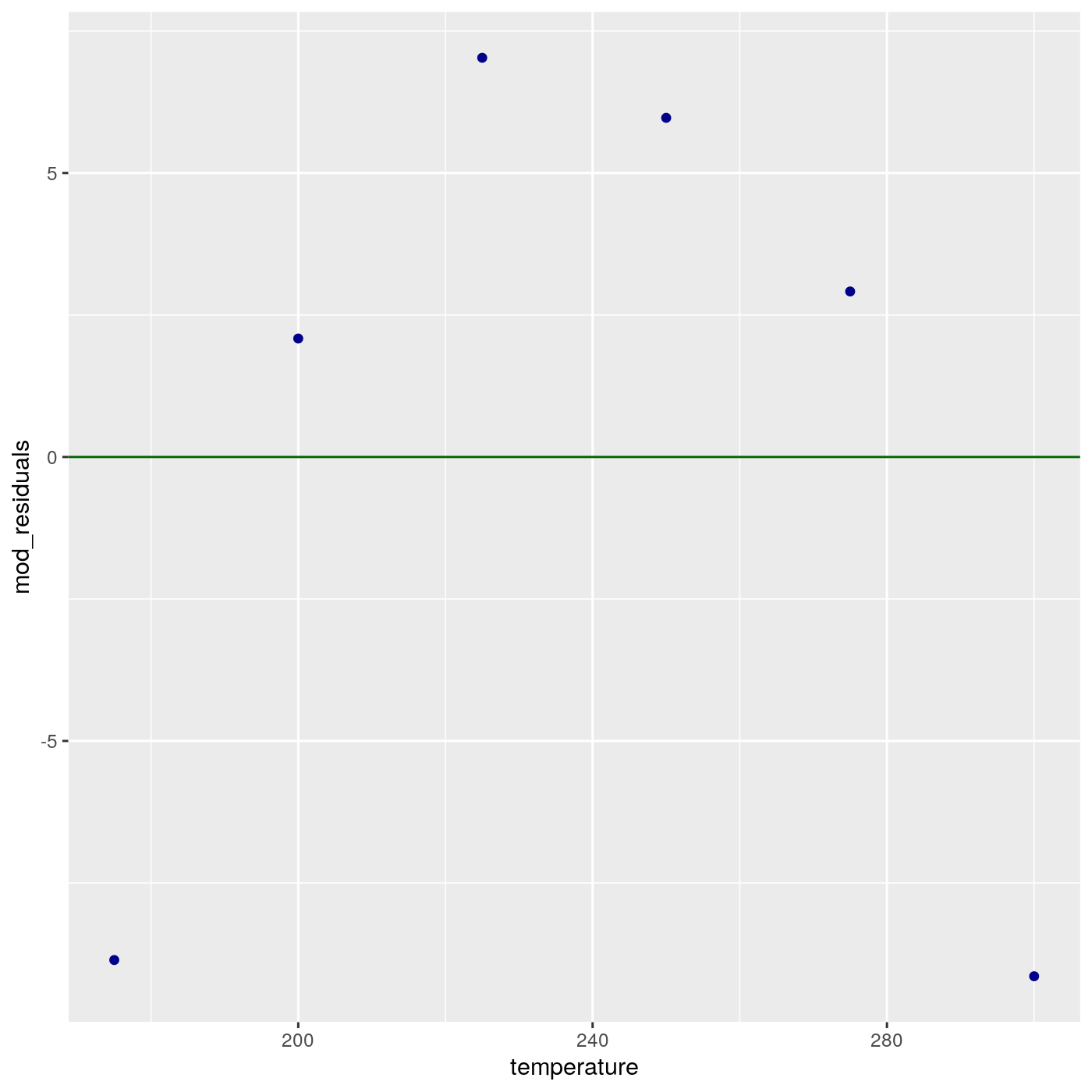
Figura 18.6: Scatterplot dei residui Vs. temperatura
Si nota una chiara curvatura.
A questo punto quindi possiamo provare un modello quadratico:
o, alternativamente:
Oa guardiamo il riepilogo del modello
##
## Call:
## lm(formula = conversion ~ temperature + I(temperature^2), data = polyester)
##
## Residuals:
## 1 2 3 4 5 6
## -0.10714 0.33571 0.02857 -1.02857 1.16429 -0.39286
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -1.888e+02 1.372e+01 -13.76 0.000830 ***
## temperature 2.197e+00 1.182e-01 18.59 0.000340 ***
## I(temperature^2) -4.200e-03 2.481e-04 -16.93 0.000449 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.9474 on 3 degrees of freedom
## Multiple R-squared: 0.9962, Adjusted R-squared: 0.9937
## F-statistic: 392.6 on 2 and 3 DF, p-value: 0.0002348Tutti i parametri sono significativi, e questo conferma l’osservazione iniziale di una relazione lineare.
18.2.5 Analisi dei residui
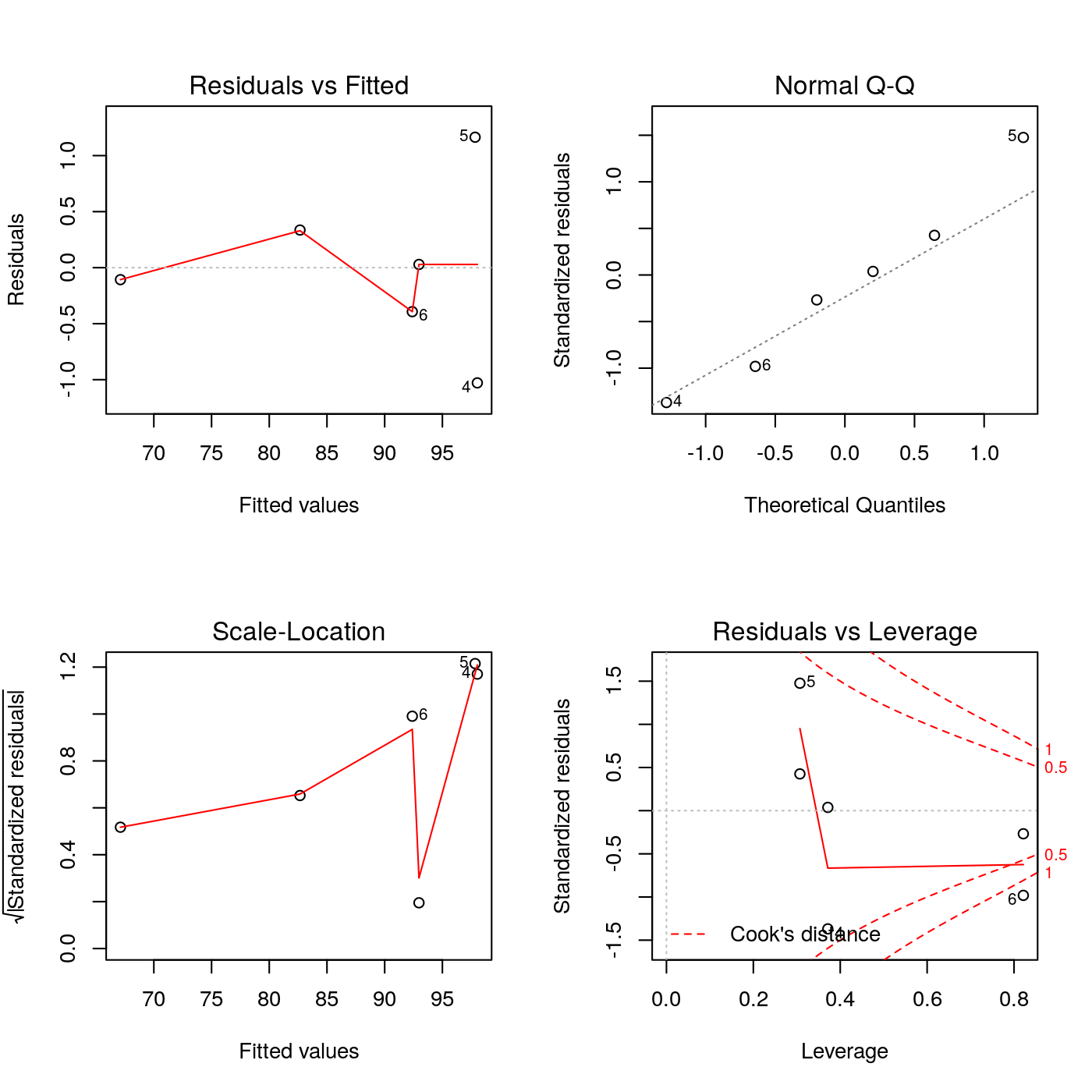
Figura 18.7: Grafico dei residui del modello finale
Il grafico dei residui sembra piuttosto “regolare”.
Come verifica aggiuntiva, possiamo provare anche l’agigunta di un termine cubico della temperatura.
##
## Call:
## lm(formula = conversion ~ temperature + I(temperature^2) + I(temperature^3),
## data = polyester)
##
## Coefficients:
## (Intercept) temperature I(temperature^2) I(temperature^3)
## -1.663e+02 1.902e+00 -2.933e-03 -1.778e-06##
## Call:
## lm(formula = conversion ~ temperature + I(temperature^2) + I(temperature^3),
## data = polyester)
##
## Residuals:
## 1 2 3 4 5 6
## -0.19048 0.45238 0.09524 -1.09524 1.04762 -0.30952
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -1.663e+02 1.168e+02 -1.424 0.290
## temperature 1.902e+00 1.524e+00 1.248 0.338
## I(temperature^2) -2.933e-03 6.519e-03 -0.450 0.697
## I(temperature^3) -1.778e-06 9.139e-06 -0.195 0.864
##
## Residual standard error: 1.15 on 2 degrees of freedom
## Multiple R-squared: 0.9963, Adjusted R-squared: 0.9907
## F-statistic: 177.8 on 3 and 2 DF, p-value: 0.005598## Analysis of Variance Table
##
## Model 1: conversion ~ temperature + I(temperature^2) + I(temperature^3)
## Model 2: conversion ~ temperature + I(temperature^2)
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 2 2.6429
## 2 3 2.6929 -1 -0.05 0.0378 0.8637Questo modello cubico non aggiunge informazione utile rispetto al modello quadratico.
Ora, usiamo il modello quadratico per producce un grafico dei valori previsti:
newdata <- data.frame(temperature = seq(min(polyester$temperature),
max(max(polyester$temperature)), length = 100))
newdata$predict <- predict(fm, newdata = newdata)
ggp <- ggplot(data=polyester, mapping = aes(x=temperature, y=conversion)) +
geom_point(colour="darkblue", size=3) +
geom_line(data=newdata, mapping=aes(x=temperature, y=predict), colour="mediumvioletred") +
xlab("temperatura (°C)") + ylab("conversione percentuale (%)")
print(ggp)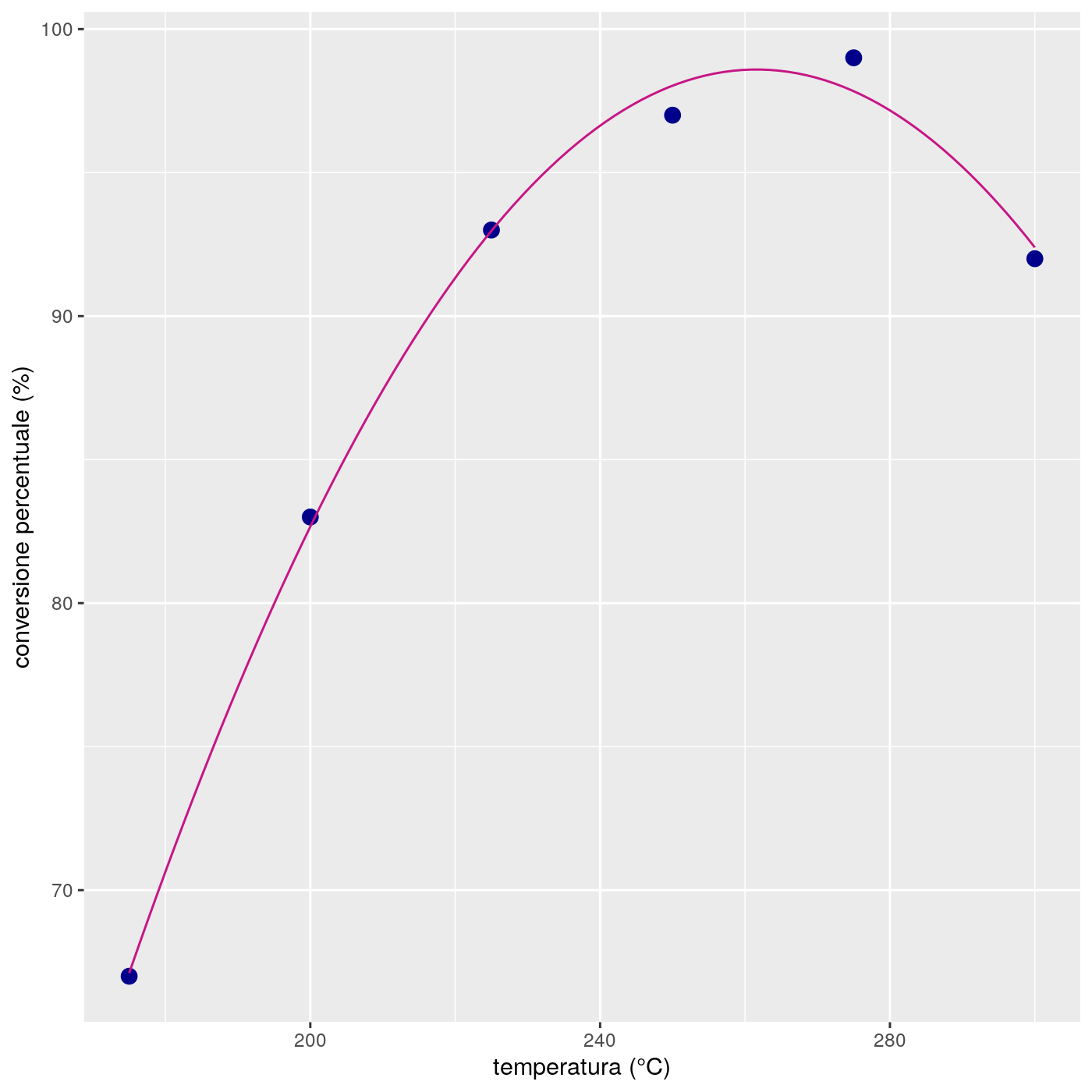
Figura 18.8: Gafico della previsione col modello quadratico